
Kleinere details uit lichtmicroscoop
Met een paar vernuftige trucs slagen wetenschappers van onder meer de Technische Universiteit Delft erin om een lichtmicroscoop details van 3 nm in beeld te laten brengen. Daarmee wordt het mogelijk om onder meer virussen en grote (eiwit)moleculen af te beelden, een wens van biomedisch onderzoekers.
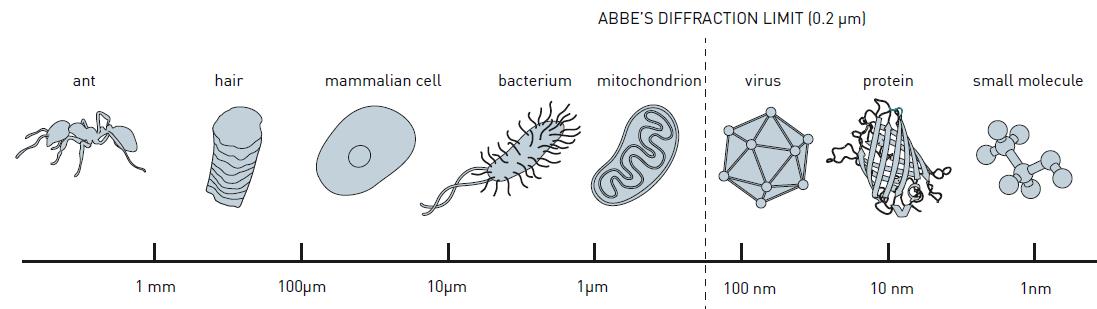
De microscoop, ooit uitgevonden door de Nederlander Antoni van Leeuwenhoek, liep op een gegeven moment tegen een grens aan: de diffractielimiet. Dit wil zeggen dat het optische instrument geen details kan afbeelden die kleiner zijn dan de halve golflengte van het gebruikte licht. Bij zichtbaar licht zit deze limiet rond de 200 – 400 nm.
Hoewel dit lange tijd een harde grens leek, slaagden onderzoekers er met trucs toch in om deze barrière te doorbreken. In 2014 kreeg een drietal wetenschappers de Nobelprijs voor de Scheikunde voor hun werk aan zogeheten superresolutie-fluorescentiemicroscopie (pdf-bestand). Met een hoop kunst en vliegwerk kregen ze de grens omlaag naar details van 20 nm.
Lichtgevende labeltjes
Hoe kun je voorbij die limiet gaan? Door delen van een af te beelden eiwit of materiaal fluorescent te maken, zodat ze licht uitstralen. Dit zwakke signaal kun je vervolgens opvangen met een lichtmicroscoop. Het probleem is alleen dat je niet alle onderdelen van de lichtgevende labeltjes kunt voorzien. Vaak maar 30 tot 50 %. Zo kun je bijvoorbeeld niet een geheel eiwitmolecuul afbeelden (een nogal lang molecuul), een droom van biomedisch onderzoekers.
Daar hebben onderzoekers van de TU Delft en collega’s uit Duitsland en de Verenigde Staten nu iets op gevonden. Zij hebben een methode verbeterd, waarbij meerdere opnames van identieke - soms honderden - objecten (een molecuul, of een onderdeel van een cel) over elkaar heen worden gelegd. Doordat van elke opname steeds een ander deel van het object oplicht, krijg je uiteindelijk toch een goed beeld. Ze publiceerden er gisteren over in het wetenschappelijke vakblad Nature Methods.
Grafische kaarten
Het verwerken van die honderden – en soms duizenden – beelden kost wel een hoop rekenkracht. Met een gewone computer zou dat dagen rekenen kosten; dankzij grafische kaarten uit de gamesindustrie en de bijbehorende speciale algoritmes lukte het om dit terug te brengen naar een paar uur.
Met het onderzoek brengen de Delftenaren lichtmicroscopie een stapje dichter bij de prestaties van elektronenmicroscopie. ‘Dat is vooral belangrijk, omdat elektronenmicroscopie tien keer zo duur is en omdat ons nieuwe proces in potentie veel sneller is dan met een elektronenmicroscoop.’ Het team heeft een gereedschap ontwikkeld dat voor het afbeelden van onderdelen van cellen, bacteriën en virussen een aanvulling kan zijn op de elektronenmicroscoop.
Nu werken de Delftse onderzoekers om nog kleinere details in beeld te brengen. De grens van 1 nm moet haalbaar zijn, verwachten ze.
Deze video illustreert de trucs waardoor een lichtmicroscoop steeds kleinere details kan laten zien:
Openingsillustratie: deze tekening laat het principe zien van superresolutie-fluorescentiemicroscopie. Vele verschillende opnames met steeds een paar lichtgevende punten worden achter elkaar gezet en leveren zo een behoorlijk goed beeld op van het object voor de microscoop. Dat is in dit geval de formule die aangeeft wat de onzekerheid is waarmee je een enkel molecuul kunt lokaliseren. Beeld Bernd Rieger, TU Delft
Meer artikelen

Een AI-fabriek in Groningen

Gezondheid meten via zweetdruppels
Nieuwste artikelen

Een AI-fabriek in Groningen






